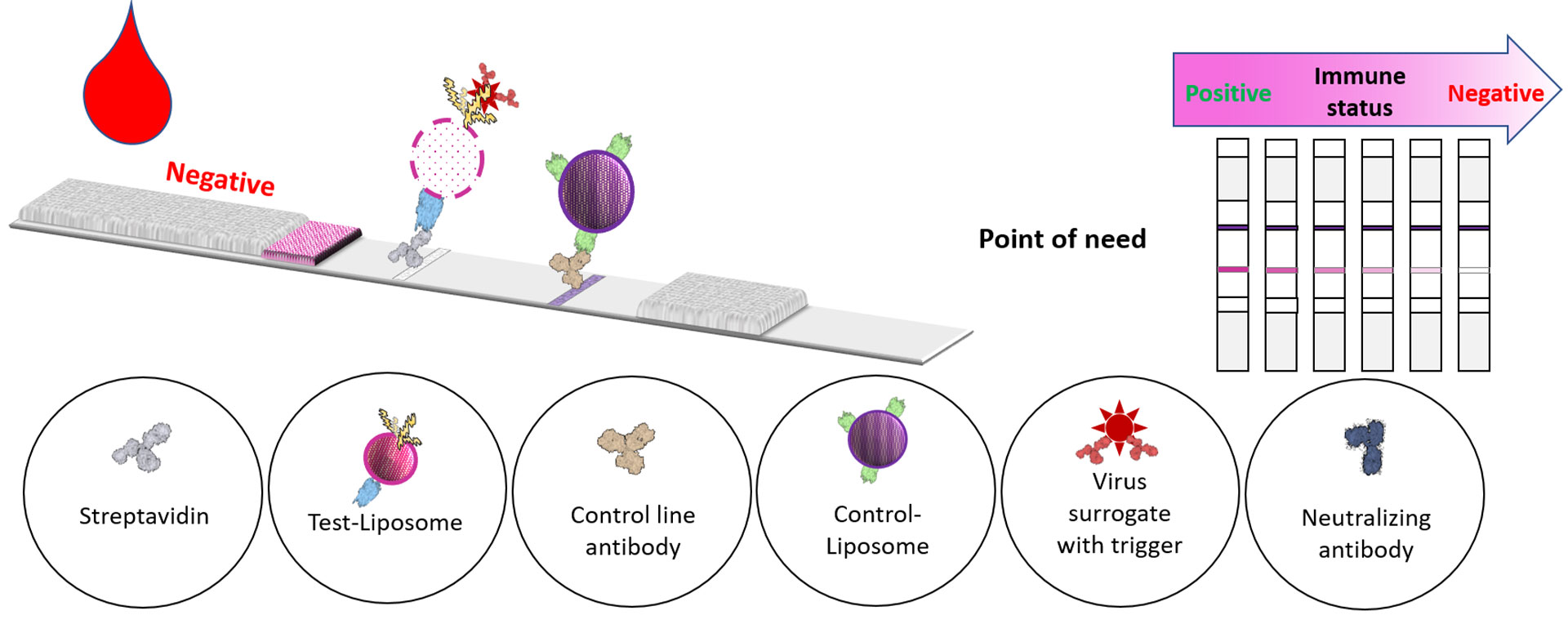
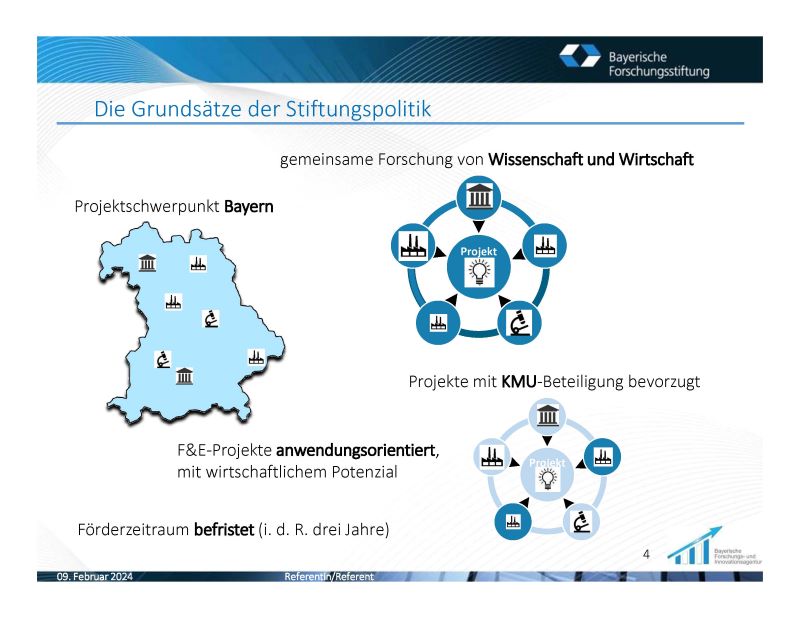
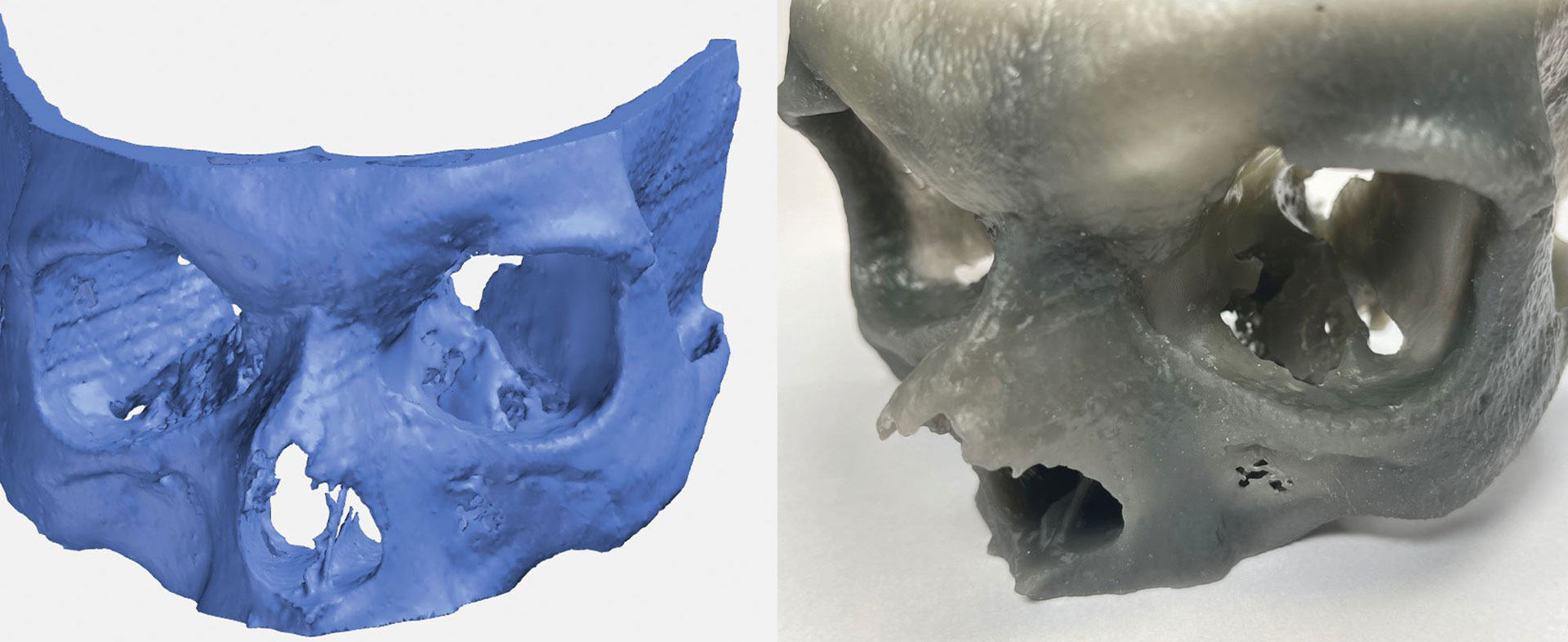
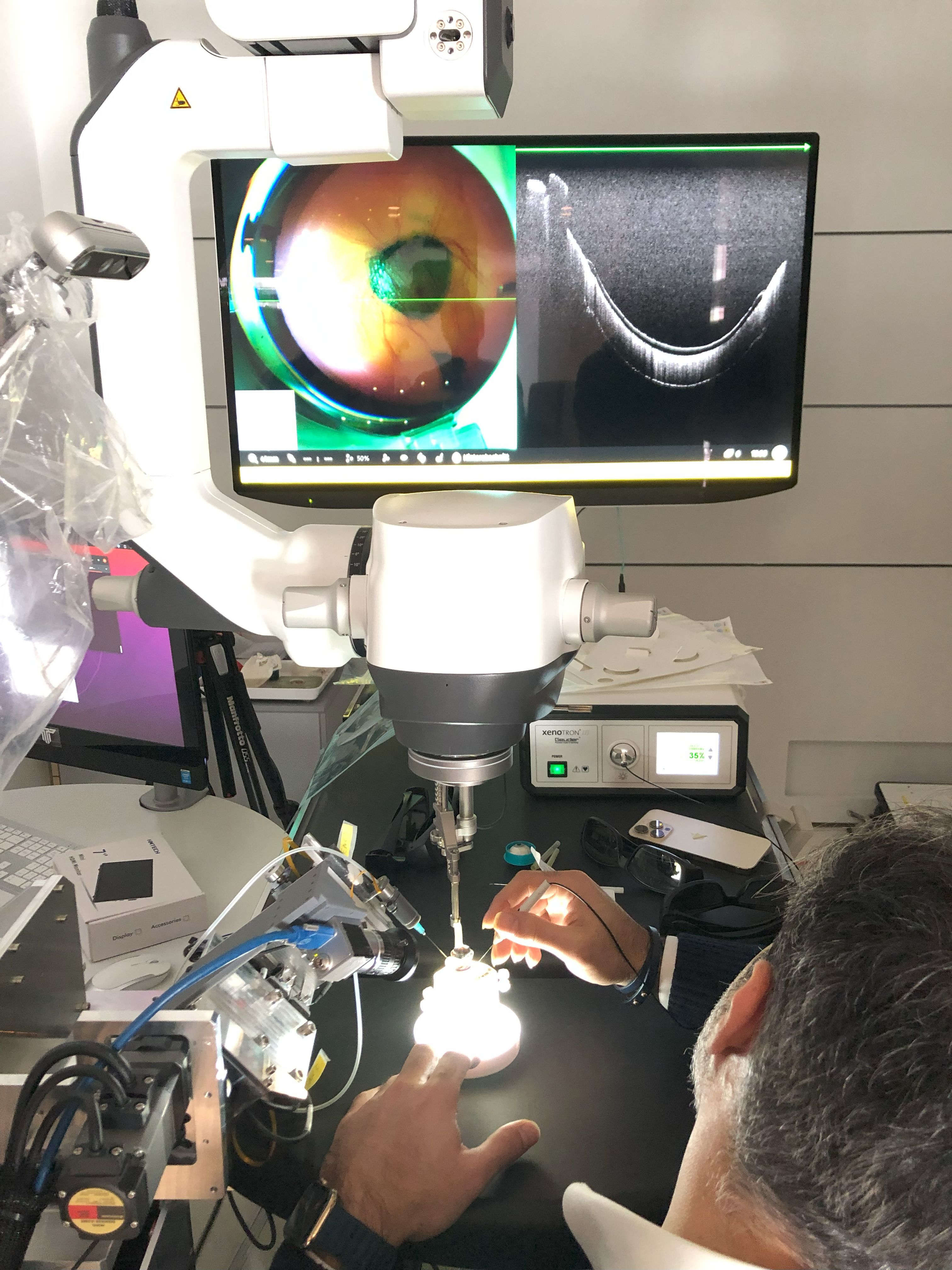
Für therapeutische Antikörper wird häufig die subkutane Injektion kleiner Volumina angestrebt. Um die erforderliche Dosis in dem begrenzten Volumen verabreichen zu können, sind hohe Konzentrationen notwendig. Dies erhöht die Gefahr, dass die Proteinmoleküle aggregieren, es zur Phasentrennung der Lösung kommt und die Viskosität zu hoch wird für die Injektion. Insgesamt kann sowohl die prozesstechnische als auch die klinische Entwicklung des Wirkstoffs beeinträchtigt werden.
Auslöser für diese Phänomene ist die intrinsische Neigung von Proteinmolekülen zur Wechselwirkung miteinander. Diese Selbstinteraktion (SI) wird wesentlich durch die Molekülstruktur und die Formulierung beeinflusst.
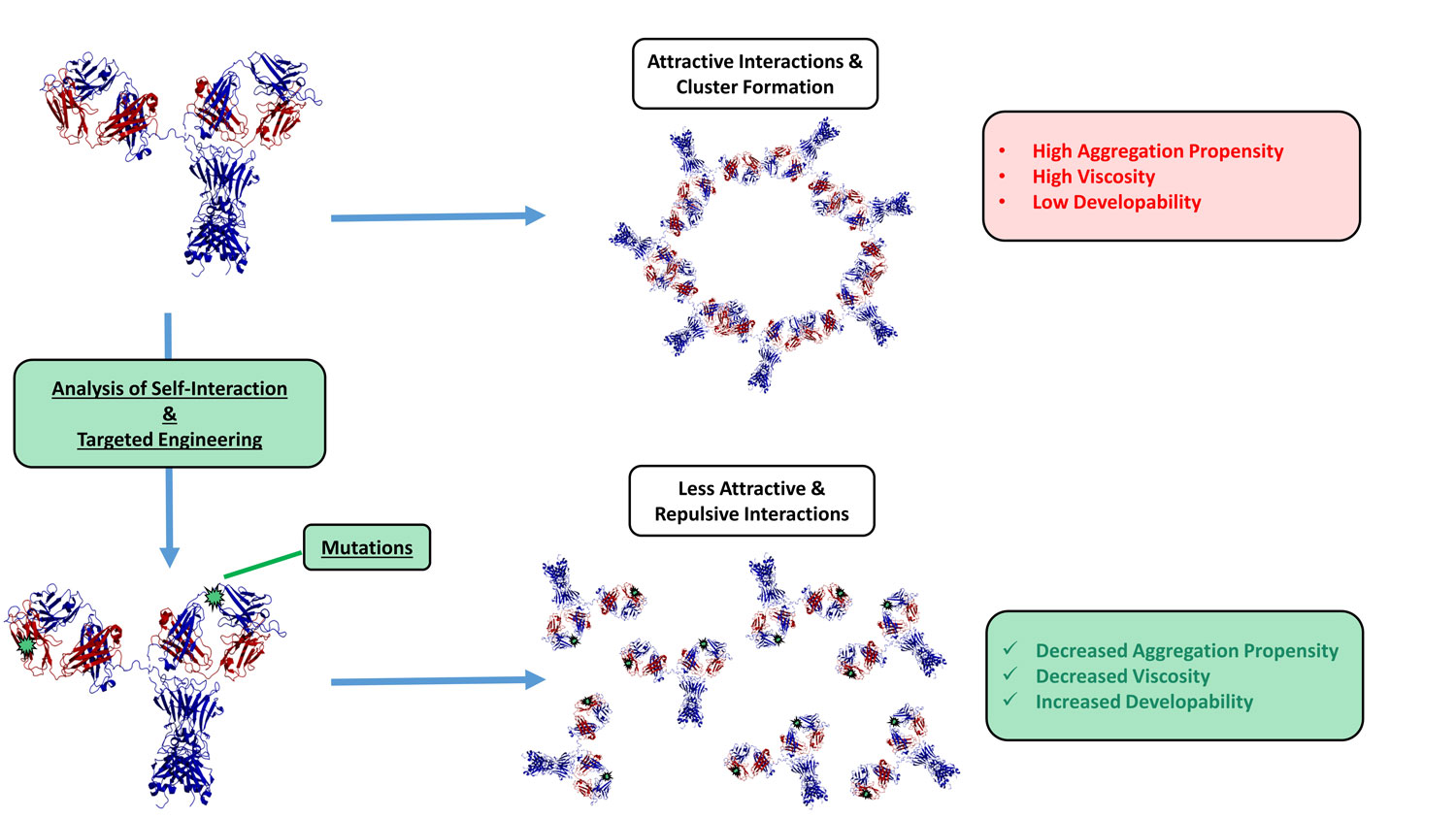
Ziel des Projekts Self-I-E war es, allgemeingültige Zusammenhänge zwischen Eigenschaften der Antikörpermoleküle wie Primärstruktur, bestimmte Sequenzmotive und die Neigung zu SI zu identifizieren sowie Wege zur Verringerung der SI mittels gezielter Modifikation der Antikörpersequenzen aufzuzeigen. Hierzu wurde die SI ausgewählter Modellmoleküle mit bereits etablierten oder — basierend auf Bio-Layer-Interferometrie und Wasserstoff-Deuterium-Austausch — im Projekt neu entwickelten komplementären Methoden analysiert und mit Computermodellierung ergänzt. Als Ursache der beobachteten SI konnten dabei hydrophobe Sequenzmotive in den Antigen-bindenden Regionen der Antikörper identifiziert werden. Durch rationale Mutagenese und das Einbringen geladener Aminosäurereste in räumliche Nähe zu den hydrophoben Bereichen gelang es, die SI und die Viskosität zu reduzieren, ohne die Antigenbindung zu beeinträchtigen.
Die Ergebnisse von Self-I-E zeigen Wege auf, um die Selbstinteraktionstendenz von Wirkstoffkandidaten bereits früh in der Forschung zu analysieren und zu verbessern und so die Entwicklungschancen zu erhöhen.