Bislang basiert die Formulierungsentwicklung für Proteintherapeutika weitgehend auf Daten zeitabhängiger Stabilitätsstudien. Dieser Ansatz ist zeit- und kostenintensiv und erfordert große Mengen an Protein. In diesem Projekt wurden daher innovative biophysikalische Methoden hinsichtlich ihrer Vorhersagekraft für die Formulierungsoptimierung in Kombination mit modernen Bioinformatik-Algorithmen evaluiert. Das Ziel ist es, zeitabhängige Stabilitätsstudien weitgehend zu ersetzen.
Mit diesen Methoden sollten geeignete biophysikalische Parameter identifiziert werden, die mit den Ergebnissen aus zeitabhängigen Stabilitätsstudien korreliert werden können. Dieses Pilotprojekt sollte damit ein zukünftiges größeres Projekt vorbereiten, in dem die Auswahl der Formulierungsbestandteile und die Formulierungsentwicklung durch Bestimmung spezifischer Kenngrößen in Verbindung mit bioinformatischen Analysen drastisch beschleunigt und optimiert werden soll.
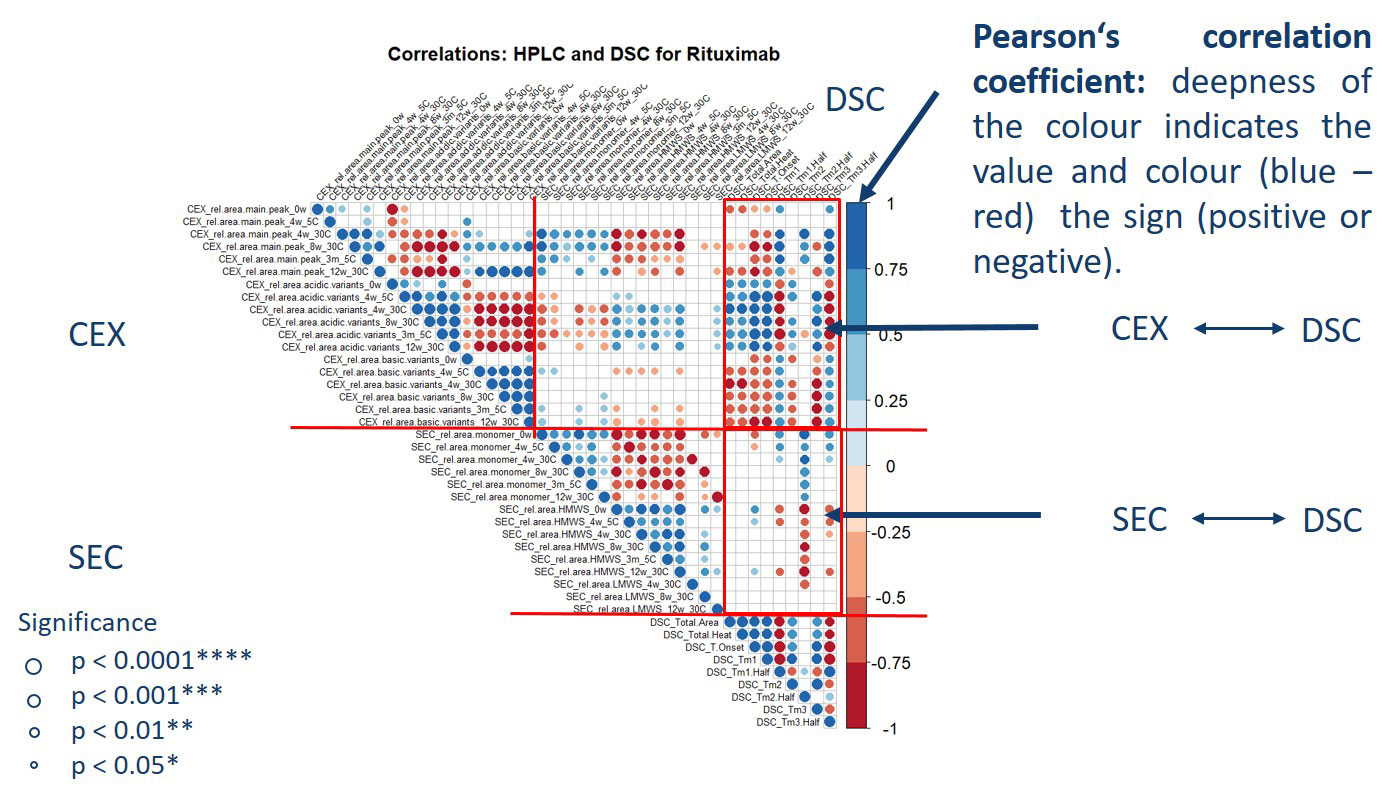
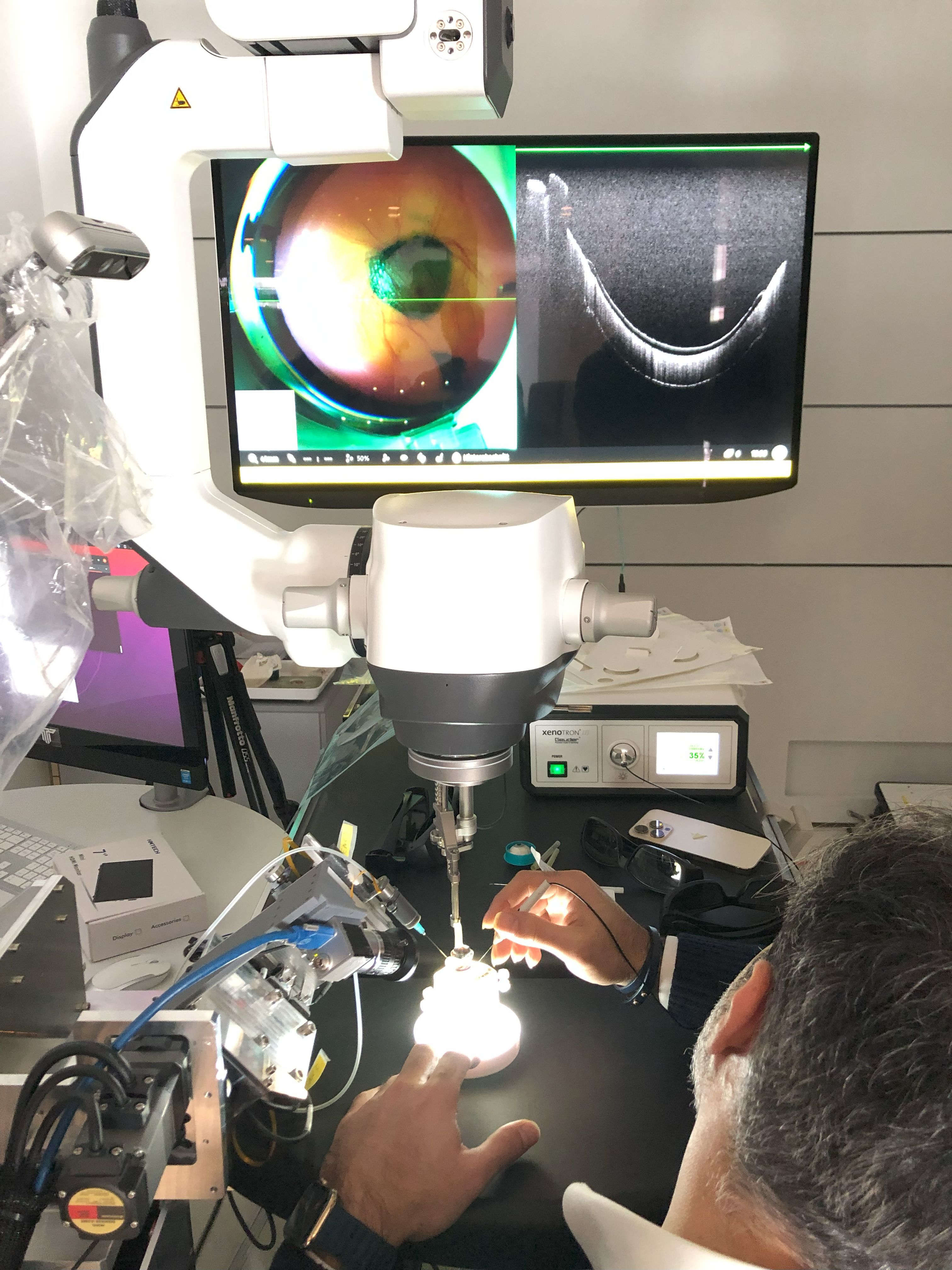
Zunächst wurden die anzuwendenden biophysikalischen Methoden zur Analyse der konformationellen sowie der kolloidalen Stabilität für zwei therapeutische Antikörper implementiert und die Abhängigkeit der Parameter von der Formulierungsumgebung analysiert. Danach wurden diese Antikörper in selektierten, sehr gut bzw. schlecht stabilisierenden Formulierungen hinsichtlich bestimmter biophysikalischer Parameter untersucht. Parallel wurden traditionelle, zeitabhängige Stabilitätsstudien durchgeführt. Die bioinformatische Analyse von Korrelationen zwischen den Ergebnissen der zeitabhängigen Studien und der biophysikalischen Parameter ergaben relevante und teilweise überraschende Korrelationen, z. B. zwischen Parametern, die die thermodynamische Stabilität beschreiben und SEC-HPLC-Parametern von zeitabhängigen Stabilitätsstudien, deren Vorhersagepotenzial in einem Folgeprojekt weiter evaluiert werden soll.