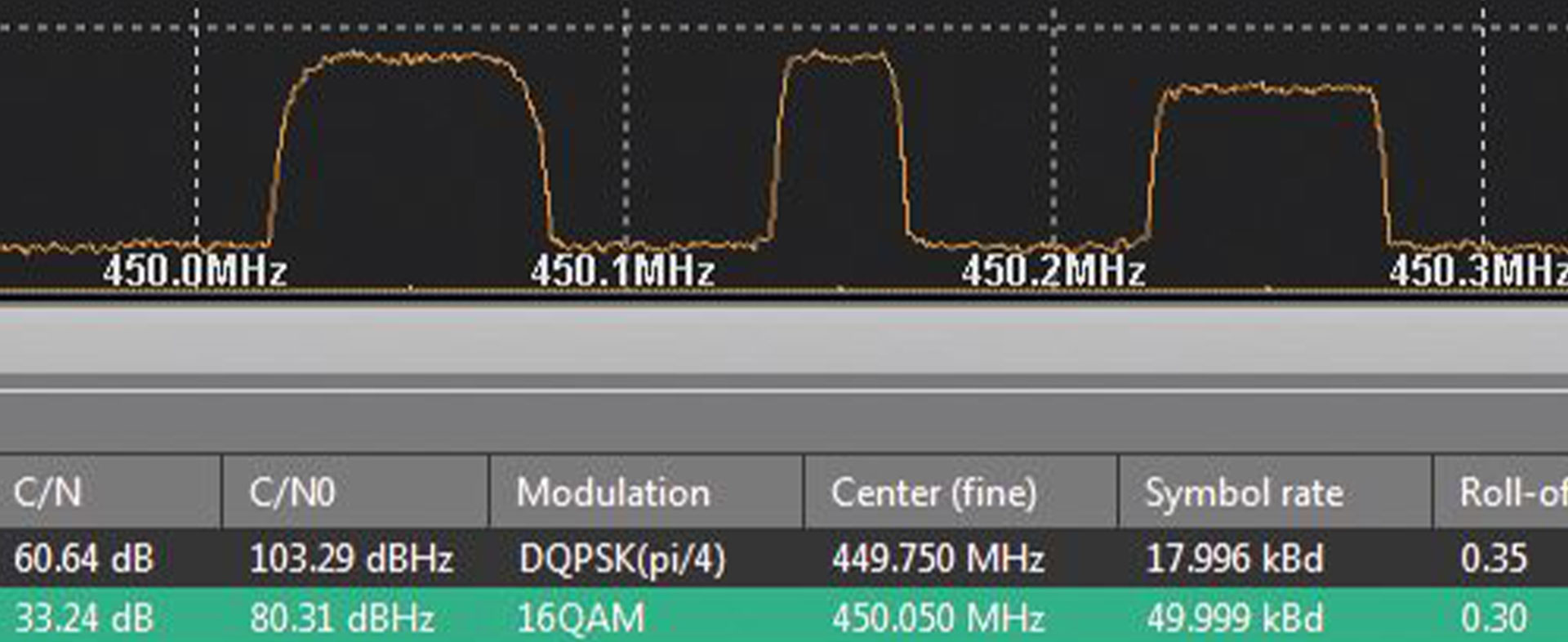
Die Entwicklung paralleler molekulardiagnostischer Tests erfordert aufwändige LaborTestreihen. Mit leistungsfähigen Bioinformatik-Anwendungen können bessere Assays günstiger entwickelt werden.
Extrem miniaturisierte DNA-Chips bieten parallele DNA-Diagnostik im Nanoformat. Sie stellen eine verheißungsvolle Technologie für einen Routinetest auf hunderte von Organismen, wie etwa bakterielle Krankheitserreger, in einem einzigen Assay dar.


rechts: DNA-Chip auf Siliziumbasis – 32 diagnostische Bereiche zum Nachweis von Nukleinsäuren (Quelle: Technische Universität München, Lehrstuhl für Rechnertechnik und Rechnerorganisation, Institut für Informatik)
Die eigentlichen diagnostischen Elemente sind dabei kurze DNA-Einzelstränge, die DNA-Sonden. Ein zentrales Problem bei der Entwicklung eines parallelen diagnostischen Assays ist die Zusammenstellung eines Satzes an DNA-Sonden, der beim Einsatz optimale Sensitivität und Spezifität garantiert. In langwierigen Labortests muss der Sondensatz derzeit hinsichtlich seines Nachweisvermögens evaluiert und durch Austausch von ungeeigneten Sonden optimiert werden. Lange Entwicklungszeiten und hohe Entwicklungskosten sind die Folge.
Im Rahmen dieses Projekts soll eine leistungsfähige Bioinformatik-Software entwickelt werden, die molekulare Signaturen in großen Genomsequenzdatenbanken aufspürt und je nach diagnostischem Anspruch und individuellem Einsatzbereich optimale DNA-Sondensätze berechnet. Um die riesigen Datenmengen effizient analysieren zu können, müssen intelligente Algorithmen entwickelt und implementiert werden. Dazu kommen die Methoden des parallelen und verteilten Rechnens zum Einsatz. Zudem werden kombinatorische Verfahren entwickelt, die Qualitätseinschätzung und -optimierung ermöglichen.
Diese Bioinformatik-Software wird praxisnah bei der Entwicklung zweier paralleler diagnostischer Assays eingesetzt, iterativ geprüft und weiterentwickelt.
Mit Hilfe umfassenderer Sequenzsuchverfahren und genauerer In-silico-Evaluierung kann ein weitaus größerer Teil der Arbeit als bisher vom Labor auf den Computer verlagert werden. Infolgedessen können präzisere parallele diagnostische Assays schneller und deutlich billiger entwickelt werden.