Im Rahmen des Projektes sollten spezifische Sequenzen zum Nachweis des hochpathogenen Clostridium difficile Ribotyp 027 ermittelt und für ein routinetaugliches Diagnostikum genutzt werden. Ein Nachweissystems für den C. difficile RT 001 gehörte ebenfalls zu den Projektzielen.
Auf miniaturisierten DNA-Chips und diagnostischen DNA-Sonden basierende Diagnostikverfahren zu hochparallelem Nachweis und schneller Identifizierung von Mikroorganismen und Viren – insbesondere im klinischen Bereich – konnten zu Beginn der Projektplanung als zukunftsweisende Technologie angesehen werden.

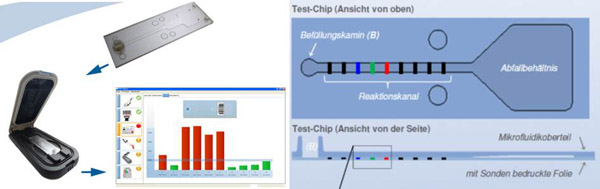
rechts: Abb.2: Prinzip und den Aufbau des „POC“-Systems
Die Herausforderungen betrafen die Bereitstellung von leistungsfähiger und benutzerfreundlicher Bioinformatik-Software zur effizienten Analyse der permanent rapide anwachsenden (Genom)-Sequenzdatenflut hinsichtlich Auffindung und Beurteilung diagnostischer Signaturen (Sonden), die begleitende und ergänzende Evaluierung der Signaturen sowie Adaptierung der Sonden für experimentelle Verfahren im mikrobiologischen Labor und letztlich deren Anpassung für praxisorientierte kommerzialisierbare Diagnosekits.
Im Bereich der Bioinformatik wurden computerbasierte Lösungen und Softwareapplikationen für die schnelle Beschaffung von großen Gen- und Genom-Sequenzdatensätzen, deren lokaler Verwaltung und kompletter Indizierung für schnelle und umfassende Signatursuchen zum Primer- und Sondendesign entwickelt und etabliert. Im Bereich der Mikrobiologie wurden die bioinformatisch gewonnenen Daten durch experimentell erhobene Befunde ergänzt und zur Entwicklung und Evaluierung von Nachweisverfahren mit Schwerpunkt auf klinisch bzw. hygienisch relevanten Organismen verwendet. Zur Praxistauglichkeit wurden Assays zur Identifizierung und Differenzierung von high- und low-risk Papillomviren (HPV) sowie Clostridium difficile Stämmen unterschiedlich aggressiver Pathogenität weiter entwickelt. Ein Clostridium-difficile-Nachweiskit konnte zur Marktreife gebracht werden.